와인품질 예측 모델링_선형회귀분석, 부분회귀플롯
이곳에서 와인속성이 담긴 red, white 와인 데이터를 다운받는다.

원본에서 ;을 열 구분자로 사용하여 엑셀로 바로 읽으면 이렇게 된다.
# 데이터 불러오기
import pandas as pd
red_df = pd.read_csv('C:\doit\winequality-red.csv', sep = ';', header = 0, engine = 'python')
white_df = pd.read_csv('C:\doit\winequality-white.csv', sep = ';', header = 0, engine = 'python')
red_df.to_csv('C:\doit\winequality-red2.csv', index = False)
white_df.to_csv('C:\doit\winequality-white2.csv', index = False)
# 첫번째 열에 컬럼 추가하고 값 넣기
red_df.insert(0, column = 'type', value = 'red')
red_df.head()
white_df.insert(0, column = 'type', value = 'white')
white_df.head()레드, 화이트 구분할 수 있는 type 컬럼을 삽입하고 각 데이터프레임을 concat() 사용하여 하나의 데이터프레임으로 합친다.
# 파일 결합하기
wine = pd.concat([red_df, white_df])
print(wine.shape) # 행, 열의 개수 확인
wine.to_csv('C:\doit\wine.csv', index = False)# 기본정보 확인하기
wine.info()
# 열 이름 공백에 _으로 치환하기
wine.columns = wine.columns.str.replace(' ', '_')
wine.head()컬럼 이름 작성 시 오타가 날 수 있으니 컬럼 이름에 공백을 _로 치환한다.
선형회귀분석모델을 만들기 위해 다음과 같은 절차를 거친다.
2-1. 와인 데이터 기술 통계 구하기
2-2. 각 와인 그룹 품질에 대한 t-검정 수행하기
2-3. 와인속성을 독립변수, 등급을 종속변수로 선형회귀분석모델 만들기
2-1. 기술통계 구하기
#기본정보 확인
wine.describe()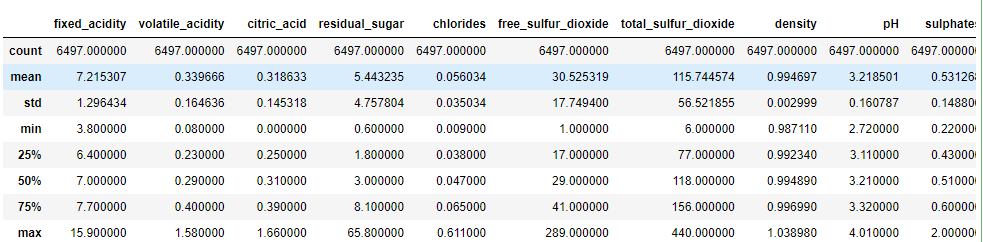
wine.quality.value_counts()
#type별 그룹화로 기술통계 구하기
wine.groupby('type')['quality'].agg(['mean', 'std', 'max'])
2-2. t검정
# t검정 위한 scipy 패키지의 stats 함수/ 회귀분석에 필요한 statsmodels.formula.api 패키지의 ols, glm 함수
from scipy import stats
from statsmodels.formula.api import ols, glmred_quality = wine.loc[wine['type'] == 'red', 'quality'] # type이 red인 행과 quality 열만 뽑
white_quality = wine.loc[wine['type'] == 'white', 'quality']# type이 white인 행과 quality 열만 뽑# scipy 패키지의 stats.ttest_ind() 사용하여 t 검정하고 그룹 차이 확인하기
stats.ttest_ind(red_quality, white_quality, equal_var = False)
t검정 결과가 유의하면 해당 회귀계수는 독립변수와 종속변수 간에 상관관계가 있다는 것을 의미한다. 이경우 p-value가 0.05보다 작으므로 회귀계수가 유의하다고 판단할 수 있다.
2-3. 선형회귀분석 수행
와인 속성들이 와인품질에 어떠한 영향을 미치는지 알기위해 선형회귀 분석을 사용한다.
종속변수는 quality, 독립변수는 종속변수와 type을 제외한 나머지 속성으로한다. (회귀식 독립변수는 연속형이여야 하기 때문)
어떤 속성들이 품질과 관련이 있는지, 어느정도 상관관계가 있는지 알 수 있다.
#선형회귀모델: statsmodel.formula.api의 ols() 사용
Rformula = 'quality~ fixed_acidity + volatile_acidity + citric_acid + residual_sugar +\
chlorides + free_sulfur_dioxide + total_sulfur_dioxide +\
density + pH + sulphates + alcohol'
reg_result = ols(Rformula, data = wine).fit() #ols(종속변수~ 독립변수), fit() 실행
reg_result.summary()OLS 선형회귀모델을 fit()을 사용하여 실행한다.

이 데이터에 따르면 density 회귀계수가 -54.9669로, 밀도가 낮을수록 와인품질이 좋다고 할 수 있겠다.
3. 예측
이제 만든 생성된 모델로 예측을 해보고 실제 등급과 비교해본다.
# quality와 tyoe 컬럼 제외 추출
sample = wine.drop(columns = {'quality', 'type'})
sample = sample[0:5][:] #0부터 4행까지 5개 샘플만 추출
sample_predict = reg_result.predict(sample)
sample_predict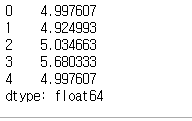
#예측한 품질 실제 품질 비교하기
wine[0:5]['quality']
다음은 새로운 속성값을 넣어 등급 예측을 해본다.
# 임의로 넣어 예측해보기
data = {'fixed_acidity' : [8.5, 8.1], 'volatile_acidity':[0.8, 0.5],
'citric_acid':[0.3, 0.4], 'residual_sugar':[6.1, 5.8], 'chlorides':[0.055, 0.04],
'free_sulfur_dioxide':[30.0, 31.0], 'total_sulfur_dioxide':[98.0, 99],
'density':[0.996, 0.91], 'pH':[3.25, 3.01], 'sulphates':[0.4, 0.35], 'alcohol':[9.0, 0.88]}
sample2 = pd.DataFrame(data, columns = sample.columns)
sample2
예측값을 새로운 컬럼에 포함시켜본다.
sample2_predict = reg_result.predict(sample2)
sample2['q_predict'] = sample2_predict
sample2
다음은 분석결과를 시각화한다.
4-1. 타입에 따른 품질등급 히스토그램 그리기
# 히스토그램으로 나타내기
import matplotlib.pyplot as plt
import seaborn as sns
sns.set_style('dark')
sns.distplot(red_quality, kde = True, color = 'red',
label = 'red wine')
sns.distplot(white_quality, kde = True,
label = 'white wine')
plt.title('Quality of wine type')
plt.legend() #차트범례 설정
plt.show()
x축 quality, y축 확률밀도 함수값.
4-2. 부분회귀 플롯으로 시각화하기
선형회귀분석을 이용해서 각 독립변수의 부분회귀 플롯을 구한다.
# 부분회귀 플롯
# 하나의 독립변수가 종속변수에 미치는 영향력을 각 시각화하기
import statsmodels.api as sm
others = list(set(wine.columns).difference(set(["quality", "fixed_acidity"])))
p, resides = sm.graphics.plot_partregress("quality","fixed_acidity", others, data = wine, ret_coords = True)
plt.show
fig = plt.figure(figsize = (8, 13))
sm.graphics.plot_partregress_grid(reg_result, fig = fig)
plt.show()


부분회귀플롯이란, 각 독립변수들을 하나씩 고정하고 나머지 독립변수들을 제외한 회귀모델을 그리는 것을 말한다. 여기에서 나타나는 기울기는 하나의 독립변수가 종속변수에 미치는 영향을 나타내는거다.
끝_!